作为一种具有“推动者”(Enabler)地位的先进技术,人工智能在各大科学领域都有着极大的应用潜力,尤其是生物医疗领域。近日,麻省理工学院的研究人员通过机器学习模型分析氨基酸序列,在无需其他任何蛋白质结构信息作为输入数据下,成功的掌握了蛋白质的三维结构,并进一步的预测其生物学功能。
这项研究将在今年 5 月的国际学习表征会议(ICLR)上发表,相关论文目前正在收集评论,其成果有望改进现有的蛋白质结构预测手段,帮助科学家设计和测试新蛋白质,用于药物研发和生物研究等目的。
蛋白质是由氨基酸组成的线性链,且每一种都有自己独特的氨基酸序列,通过肽键连接。根据氨基酸的序列和物理相互作用,蛋白质分子可以折叠成非常复杂的三维结构,而这种结构决定了其生物学功能,比如对药物产生什么样的反应。
图 | 蛋白质分子的三维模型 (来源:MIT News)
然而,经过数十年的研究和多种成像技术的发明,人们仍然只掌握了很少一部分蛋白质的结构——在已知的上百万种蛋白质中,我们只了解其中几万个的结构。
鉴于氨基酸序列的信息相对比较容易获取,麻省理工学院的研究人员想到了擅长预测和寻找规律的机器学习技术,希望可以组合不同的氨基酸序列,从而找到新的蛋白质结构。这是一个富有挑战性的任务,因为不同的氨基酸序列可以形成非常相似的结构,而且没有很多结构可以用来训练模型。
研究的第一作者 Tristan Bepler 认为,类似的研究可以将蛋白质结构的预测边缘化,因为只需要氨基酸序列,就可以推测出蛋白质的功能。
预测蛋白质结构
研究团队没有直接基于蛋白质结构建立预测模型,而是首先尝试编码蛋白质的结构信息,将其变为一种易于计算的表达方式,再训练模型学习特定氨基酸的功能,找出不同蛋白质结构之间的相似度,然后用这一数据来监督模型。
依据蛋白质结构分类数据库(SCOP)的数据,研究人员对大约 22,000 种蛋白质进行了模型训练,通过其结构和氨基酸序列的相似性分成不同类别。
他们随后将蛋白质结构和氨基酸序列编码,转换成套嵌(Embedding)的数字表达形式,以随机配对的形式放入预测模型中,每组套嵌包括两个氨基酸序列的相似性信息,经过对比和计算,可以得出蛋白质三维结构的相似度,最后根据其中每个氨基酸的位置和接触来预测其功能。
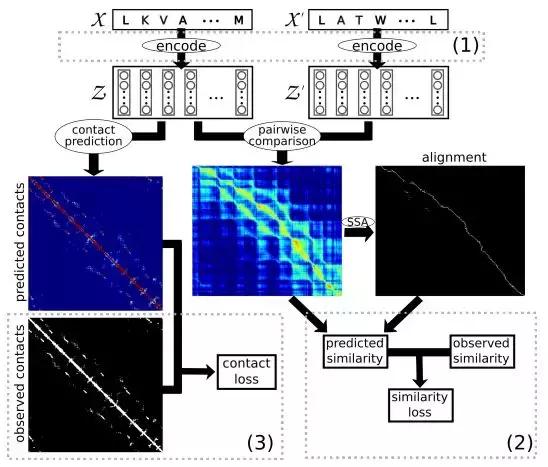
图 | 模型的工作过程 (来源:Tristan Bepler)
我们可以用类比搞清楚它的运作机制:在自然语言处理(NLP)的语义分析中,也存在类似的套嵌概念,比如两套单词的套嵌(值)越相近,它们在句子中一起出现的可能性就越大。
类比到蛋白质结构预测模型中,蛋白质就像“句子”,氨基酸就像“单词”,那么氨基酸序列也就成为了“单词的组合形式”,而套嵌则是“单词组合的相似度”,可以对比得出“句子(蛋白质)的相似度”。如果两组氨基酸序列越相近,它们的套嵌(值)就越相近,就说明两种蛋白质结构越相似。
在训练过程中,机器学习模型负责计算两组套嵌的相似性得分,然后预测出蛋白质三维结构的相似性,再与实际 SCOP 相似性得分进行比较。如果两者非常接近,就说明模型的方法没问题,反之则需要调整。
此外,该模型还会预测每组套嵌的“接触图”(Contact Map),即三维结构中每个氨基酸与其他氨基酸的距离。这有助于模型掌握氨基酸在蛋白质结构中的确切位置,从而进一步预测每种氨基酸的功能。同样的,它会跟 SCOP 数据中已知的接触图进行比较,验证判断是否准确。
作为测试,研究人员利用该模型预测哪些蛋白质可以穿过细胞膜,在仅给出一个氨基酸序列的情况下,所得结果的准确率已经超过了现有的最先进的模型。
接下来,研究团队的目标是将该模型应用于更多预测任务,拓展到更广泛的蛋白质工程领域,例如检测破坏蛋白质结构的有害突变,以及确定哪些氨基酸序列片段可以与小分子结合,这对于药物研发至关重要。该模型还可以用于蛋白质设计领域,通过对套嵌等数据的分析,可以找出更多使蛋白质发光的波长。
“机器学习模型可以有效利用已知的氨基酸序列,分析蛋白质折叠的奥秘,进而推测未知的蛋白质结构和功能。我们的终极目标是,更有效地赋能数据驱动的蛋白质设计工程。” Bepler 强调。


 添加微信免费咨询
添加微信免费咨询
